伊成器课题组开发胞嘧啶碱基编辑器全基因组脱靶效应检测的新工具
2021年6月8日,北大-清华生命科学联合中心、北京大学生命科学学院、蛋白质与植物基因研究国家重点实验室伊成器课题组在Nature Methods杂志发表了题为“Detect-seq reveals out-of-protospacer editing and target-strand editing by cytosine base editors”的封面文章。

在本研究工作的主要贡献如下:
1.建立了一种体内无偏好性的CBE脱靶检测技术
作者通过捕获CBEADDIN EN.CITEADDIN EN.CITE.DATA[1]工具产生的中间体(dU),并进行一系列标记和富集ADDIN EN.CITEADDIN EN.CITE.DATA[2, 3]、建库,获得具有tandom C-to-T信号的脱靶位点的技术,并将其命名为Detect-seq(dU-detection enabled by C to T transition during sequencing)技术,该技术的主要技术路线如图1所示。
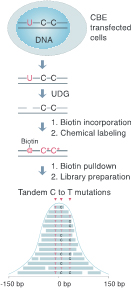
图- 1 Detect-seq技术路线图
2.通过Detect-seq技术发现CBE工具导致的全基因组上的大量脱靶位点
研究人员首先在人胚肾细胞HEK293T和人乳腺癌细胞MCF-7转染BE4maxADDIN EN.CITEADDIN EN.CITE.DATA[4],分别使用3种、2种不同的sgRNA对基因组靶向位点进行C-to-T的编辑;同时对这些样本中进行了Detect-seq检测。通过Detect-seq检测发现,除RNF2位点外,其它靶向位点均可以产生几十至数百个Cas9依赖型的脱靶位点(图2)。
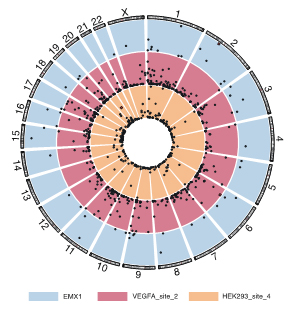
图-2在三种不同sgRNA样品中鉴定到的Cas依赖型的脱靶位点 其中蓝色圆圈代表脱靶位点(off-targets),红色方块代表靶向位点(on-target)EMX1 n=48,VEGFA_site_2 n=511,HEK293_site_4 n=245
3.发现了两种新型脱靶位点(out-of-protospacer edit和target-strand edit)
有趣的是,研究人员基于Detect-seq技术还发现了两种新型的Cas依赖型的脱靶编辑:sgRNA结合区域外编辑(out-of-protospacer edit)及靶向链编辑(target-strand edit)(图3)。sgRNA结合区域外编辑即C-to-T的编辑发生在sgRNA结合区域外;而靶向链编辑则是C-to-T编辑发生在sgRNA与DNA结合链。根据Detect-seq的结果,上述两种编辑普遍存在于Cas依赖型的脱靶位点的结合区域。
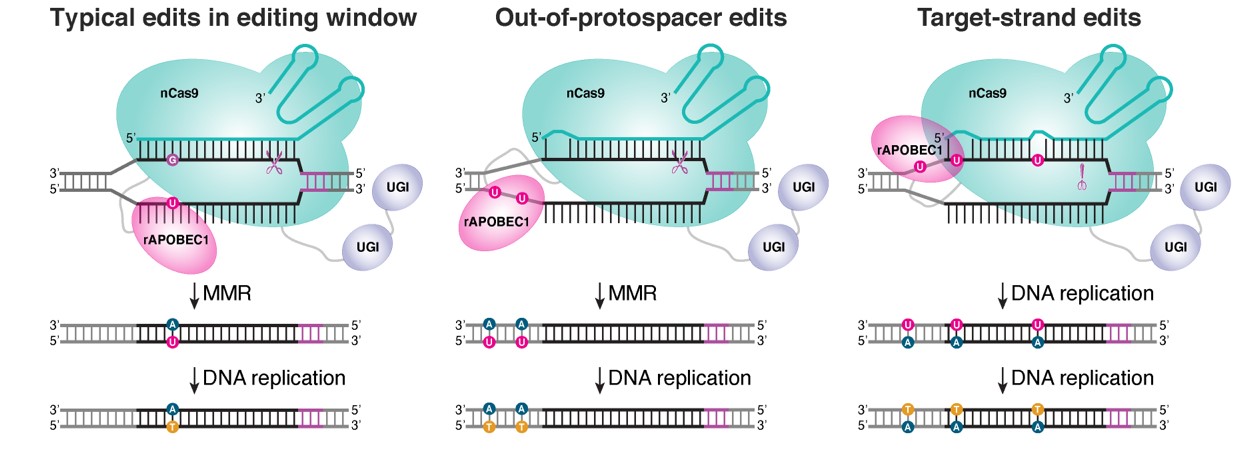
图- 3 sgRNA结合区域外编辑与靶向链编辑模式图
综上所述,Detect-seq技术为碱基编辑领域内提供了一种能够高灵敏、高特异、无偏好性地检测细胞内的CBE脱靶位点的方法。脱靶效应理解的加深,也将为阐明CBE的催化机制、优化改造更安全的CBE工具带来新的视角与可能。
北大-清华生命科学联合中心、北京大学生命科学学院伊成器教授为本文通讯作者;前沿交叉学科研究院雷芷芯博士,生命科学学院博士研究生孟浩巍、吕志聪为本文的共同第一作者;该研究得到了国家重点研发计划、国家自然科学基金以及生命科学联合中心的资助。生命科学学院高性能计算平台为该研究提供了计算资源。
原文链接:https://www.nature.com/articles/s41592-021-01172-w
博士后招聘:本实验室依托北大-清华生命科学联合中心和北京大学生命科学学院,通过化学生物学、单细胞组学、基因编辑等手段,对DNA和RNA修饰的组学技术、生物功能及其调控机制进行研究,尤其关注DNA/RNA生物学在人类疾病诊断与治疗中的应用。现诚聘博士后2-3名。详细信息请见链接:
http://www.bio.pku.edu.cn/homes/Index/news_cont/37/15652.html
参考文献
1. Komor, A.C., et al.,Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage.Nature, 2016.533(7603): p. 420-4.
2. Xia, B., et al.,Bisulfite-free, base-resolution analysis of 5-formylcytosine at the genome scale.Nat Methods, 2015.12(11): p. 1047-50.
3. Zhu, C., et al.,Single-Cell 5-Formylcytosine Landscapes of Mammalian Early Embryos and ESCs at Single-Base Resolution.Cell Stem Cell, 2017.20(5): p. 720-731 e5.
4. Thuronyi, B.W., et al.,Continuous evolution of base editors with expanded target compatibility and improved activity.Nat Biotechnol, 2019.37(9): p. 1070-1079.
- 李晟课题组与合作者揭示保护地缓解人类对兽类行为干扰的关键作用2025.12.15
- 姚蒙研究组系统评估无脊椎动物宏条形码引物,助力eDNA生物多样性研究2025.12.12
- 朱丹萌课题组与合作者揭示PICKLE调控染色质状态与基因转录的分子机理2025.12.09
- 季雄团队提出FeaSion策略揭示RNA聚合酶磷酸化的特征调控与功能2025.12.01
- 罗述金课题组古DNA研究揭示:豹猫与人共栖3500年直至汉末,家猫经丝绸之路唐代方始传入2025.11.28
- 白洋研究员入选第三期 “新基石研究员项目”资助名单2025.11.26
- 实验室主任瞿礼嘉教授2025年新当选中国科学院院士2025.11.25
- 汪阳明团队与西湖大学卢培龙团队利用人工智能辅助蛋白质设计实现精准线粒体DNA碱基编辑2025.11.18
- 肖俊宇和高宁团队联合发表多聚抗体设计策略2025.11.07
- 赵进东课题组应邀撰写藻胆体的结构和能量传递机制长文综述2025.10.28
- 郑晓峰课题组揭示代谢酶ALDOA入核激活NF-κB信号通路驱动胰腺癌进展2025.10.23
- 陆剑课题组揭示果蝇饥饿耐受的遗传架构和性别差异2025.09.29
- 遇赫课题组揭示中国家猪的起源传播与人群互动过程2025.09.26
- 季雄团队揭示INO80/SWR复合物通过BRD2和染色质景观调控Pol II转录起始2025.09.22
- 刘启昆课题组与合作者在Current Opinion in Plant Biology发文总结植物再生领域表观遗传调控的研究进展2025.09.18
- 钟上威课题组受邀发表植物光信号传导Tansley长文综述2025.09.16
- 姚蒙课题组研发新型空气eDNA采集器高效检测动植物多样性2025.09.11
- 焦雨铃研究组克隆了小麦粒重新基因,服务高产育种2025.09.02
- 魏文胜团队与合作者联合开发新型通用型CAR-T 疗法治疗血液系统恶性肿瘤2025.08.22
- 魏文胜团队开发RNA环化新方法2025.08.12
- 汪阳明团队创新双组学技术MAPIT-seq:在单细胞水平同时绘制RNA结合蛋白作用图谱与基因表达图谱的新利器2025.08.11
- 周岳课题组揭示拟南芥PRC1介导的H2Aub loop的形成机制及功能2025.08.11
- 贺新强课题组与周岳课题组合作揭示PRC2调控维管组织模式建立机制2025.08.11
- 苏晓东课题组揭示清道夫受体CD163识别底物的分子机制2025.07.23
- 陈雪梅课题组系统阐释植物miRNA成熟与功能调控机制2025.07.23
- 苏晓东课题组与合作者揭示AAV与新型受体(AAVR2)结合的分子机制2025.07.15
- 合成植物基因组2025.07.09
- 伊成器课题组与合作者建立RNA"暗码",升级生命语言2025.07.04
- 魏文胜团队系统揭秘人类基因组中“沉默突变”的功能性“低语”2025.06.26
- 肖俊宇团队与张永辉团队合作揭示嗜乳脂蛋白激活γδ T细胞的“分子钳”机制2025.06.12
- 昌增益课题组揭示蛋白质内源性(顺式)去组装元件调控细菌细胞分裂环(Z环)的动态形成2025.06.09
- 陆剑课题组揭示uORF在动物进化与发育中发挥“翻译减震器”调控作用的新机制2025.06.09
- 张蔚课题组与合作者揭示兰花螳螂程序性体色转变的分子机制及生态学意义2025.05.30
- 2025年“全国科技活动周”和“全国科技工作者日”活动2025.05.29
- 李川昀团队与合作者开发细胞通讯分析工具STCase,揭示肿瘤微环境中空间特异性通讯事件2025.05.29
- 伊成器课题组系统阐释假尿嘧啶修饰的功能与治疗应用新范式2025.05.27
- 瞿礼嘉/钟声团队应邀撰写被子植物雌-雄相互作用保证双受精成功综述2025.05.26
- 陆剑课题组与合作者共同揭示uORFs通过精细调节CLOCK蛋白的翻译参与果蝇昼夜节律及睡眠行为的调控机制2025.05.16
- 伊成器团队发展痕量样品m6A修饰定量测序方法2025.05.08
- 白洋团队联合多位顶尖科学家系统解析根际微生物组调控水稻分蘖的功能与机制2025.04.24
- 瞿礼嘉/钟声课题组发现植物传粉过程中“未雨绸缪”的“两步授粉”备份新机制,为作物逆境下的育性优化提供新思路2025.04.15
- 曾虎课题组应邀撰写综述:空间组学技术的研究进展2025.04.06
- 张迪课题组受邀综述蛋白质L-乳酰化的研究进展2025.04.04
- 苏晓东课题组揭示短序列锚定元件AE在DNA与蛋白质结合中的重要作用2025.03.31
- 魏文胜团队实现人类肿瘤免疫调控网络的单碱基精度解析2025.03.21
- 白洋团队构建全球首个作物根际"细菌+病毒"基因组数据库2025.03.13
- 季雄团队揭示RNA聚合酶亚基RPB7偶联磷酸酶CTDP1稳定Pol II并介导转录再起始2025.03.05
- 何爱彬团队利用全景单细胞组蛋白修饰实现胚胎发育谱系追踪2025.03.04
- 赵进东课题组揭示蓝细菌藻胆体与光系统II结合的新分子机制2025.02.17
- 高歌课题组提出面向大规模异质性空间转录组学切片的表征与解析新方法2025.02.12
- 周岳课题组揭示拟南芥雄性生殖细胞发育过程中染色质三维结构的动态变化过程及其重要作用2025.02.12
- 魏文胜团队发布新一代线粒体碱基编辑器助力建立疾病动物模型2025.01.23
- 贺新强课题组揭示木质部管状分子发育的microRNA调控网络2025.01.19
- 周岳课题组在Genome Biology发文揭示了拟南芥中启动子空间调控模式和喷泉结构形成机制2025.01.02
- 高歌课题组提出人类转录调控元件建模与相关非编码变异功能解析方法2025.01.02
- 魏文胜团队利用碱基编辑器筛选绘制DNA损伤应答功能元件图谱2024.12.16
- 秦跟基课题组揭示弱光下种子萌发调控新机制2024.12.05
- 刘启昆课题组开发了全新的植物细胞谱系追踪工具2024.11.26
- 周岳课题组揭示植物首个三维基因组结构蛋白及其调控机制2024.11.22
- 周岳课题组揭示植物特有的PWWP结构域蛋白调控基因表达的分子机制2024.11.22
- 国家重点实验室陈雪梅教授获得2024年度“求是杰出科学家奖”2024.11.08
- 秦跟基课题组应邀撰写品牌综述“Tansley insight”总结TCP转录因子在细胞器、细胞和器官命运决定中的重要功能2024.10.28
- 郑晓峰课组揭示USP1-ATF4-CD98hc调控ENKTL淋巴瘤患者耐药的新机制2024.09.30
- 肖俊宇课题组揭示IgM–CD5L复合物的分子机制2024.09.30
- 陆剑课题组揭示密码子使用偏好性对翻译调控的影响2024.09.30
- 魏文胜团队实现蛋白质组中丝氨酸、苏氨酸和酪氨酸位点的功能解析2024.09.24
- 王继纵/邓兴旺课题组合作解析植物光敏色素phyB光信号转导的机制2024.09.24
- 刘君/杨雪瑞课题组合作揭示m6A-cenRNA调控癌细胞着丝粒稳态的机制2024.09.23
- 伊成器教授荣获2024年“科学探索奖”2024.08.29
- 王伟课题组报道蛋白酶体调控SG稳态抵御高温胁迫的新机制2024.08.22
- 肖俊宇团队阐明IgE 高亲和力受体FcεRI 复合物的组装机制2024.08.22
- 朱玉贤院士团队发布首个棉花基因组完整图谱,阐述棉族独特折叠胚胎形成的分子与演化机制2024.08.16
- 李晴课题组报道了滞后链核小体组装和冈崎片段成熟的协同机制2024.08.13
- 魏文胜团队报道非脱氨酶依赖的嘧啶碱基编辑器TBE2024.08.03
- 李晴研究组与合作者报道真核DNA复制体介导的亲本组蛋白表观遗传信息继承新机制2024.08.02
- 张迪课题组与合作者共同报道区分蛋白质乳酰化修饰同分异构体的新方法2024.07.22
- 国际遗传工程和生物技术中心(ICGEB)总干事Lawrence Banks教授一行访问陆剑课题组2024.06.26
- 郭强课题组与合作者揭示Synaptophysin调控突触小泡生成与功能的机制2024.06.06
- 2024年全国科技周开放活动2024.05.28
- 李磊课题组解析miR408平衡植物生长和抗旱的分子机制2024.05.16
- 李晟课题组与合作者研究揭示华北地区华北豹栖息地及其景观连通性现状2024.05.06
- 陆剑课题组揭示黑腹果蝇演化历史和环境适应机制2024.04.19
- 李川昀课题组在WIRES RNA发表从头起源新基因起源特征的综述2024.04.16
- 秦跟基课题通过构建拟南芥十二重突变体揭示雌蕊顶端命运决定的分子机制2024.04.08
- 李川昀课题组与合作者揭示结构变异编码人脑特异发育的新机制2024.04.07
- 肖俊宇课题组阐明磷酸化酶激酶PhK的组装与激活机制2024.04.01
- 陆剑课题组研发SIRSVIDE模型解析病毒进化动态2024.03.28
- 陈雪梅课题组鉴定了一个新的非典型帽子修饰RNA(NAD-capped RNA)脱帽酶,揭示了NAD+帽子修饰参与基因表达调控的新机制2024.03.18
- 祝贺瞿礼嘉教授成果入选 2023 年度“中国生命科学十大进展”2024.03.08
- 李晴、高宁及合作者揭示亲本组蛋白在DNA复制叉回收的关键分子机制2024.03.07
- 伊成器课题组开发升级版RNA编辑技术RESTART v32024.03.06
- 魏文胜课题组揭示肿瘤逃逸非HLA-I类分子依赖多效型T细胞杀伤的新机制2024.02.21
- 秦跟基课题组与合作者揭示水稻花药适时开裂的分子机制2024.02.21
- 陆剑课题组发表综述探讨新冠病毒刺突蛋白的功能演化2024.02.20
- 李磊课题组与合作者揭示巨胞饮的转录调控机制2024.02.19
- 焦雨铃课题组与合作者完成首个多细胞植物染色体的部分设计与合成2024.01.27
- 伊成器和合作者报道m1A修饰酶在调控造血干细胞衰老过程中的新机制2024.01.18
- 陆剑课题组与合作者共同揭示猴痘病毒蛋白质序列和密码子使用的分子演化规律2023.12.15
- 陆剑课题组与合作者发表综述总结动物microRNA调控的趋同和趋异演化2023.11.24
- 魏文胜团队实现人类蛋白质组中赖氨酸位点的功能解码2023.11.23